Hvad er GLM i R?
Generaliserede lineære modeller er en undergruppe af lineære regressionsmodeller og understøtter ikke-normale distributioner effektivt. For at understøtte dette anbefales det at bruge glm () -funktionen. GLM fungerer godt med en variabel, når variansen ikke er konstant og distribueres normalt. En linkfunktion er defineret til at transformere responsvariablen til den passende model. En LM-model udføres med både familien og formlen. GLM-modellen har tre nøglekomponenter kaldet tilfældig (sandsynlighed), systematisk (lineær prediktor), linkkomponent (til logit-funktion). Fordelen ved at bruge glm er, at de har modelfleksibilitet, ikke behov for konstant varians, og denne model passer til størst mulig sandsynlighedsestimering og dens forhold. I dette emne skal vi lære om GLM i R.
GLM-funktion
Syntaks: glm (formel, familie, data, vægte, delmængde, Start = null, model = SAND, metode = ””…)
Her inkluderer familietyper (inkluderer modeltyper) binomial, poisson, gaussisk, gamma, kvasi. Hver distribution udfører en anden anvendelse og kan bruges i enten klassificering og forudsigelse. Og når modellen er gaussisk, skal responsen være et reelt heltal.
Og når modellen er binomial, skal responsen være klasser med binære værdier.
Og når modellen er Poisson, skal responsen være ikke-negativ med en numerisk værdi.
Og når modellen er gamma, skal responsen være en positiv numerisk værdi.
glm.fit () - Til at passe til en model
Lrfit () - betegner logistisk regressionspasning.
opdatering () - hjælper med at opdatere en model.
anova () - det er en valgfri test.
Sådan oprettes GLM i R?
Her skal vi se, hvordan man opretter en let generaliseret lineær model med binære data ved hjælp af glm () -funktion. Og ved at fortsætte med Trees-datasættet.
eksempler
// Import af et biblioteklibrary(dplyr)
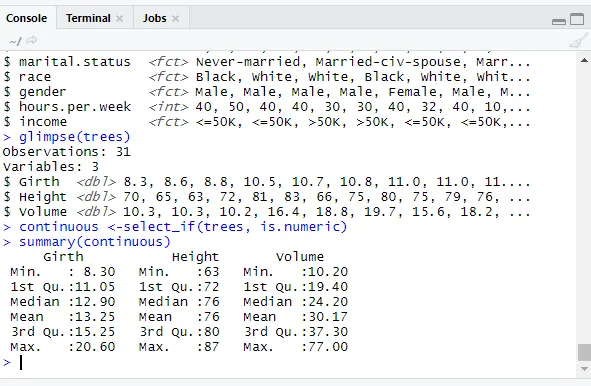
glimpse(trees)

For at se kategoriske værdier tildeles faktorer.
levels(factor(trees$Girth))
// Verificering af kontinuerlige variabler
library(dplyr)
continuous <-select_if(trees, is.numeric)
summary(continuous)
// Inklusive trædatasæt i R-søgning Pathattach (træer)
x<-glm(Volume~Height+Girth)
x
Produktion:
| Opkald: glm (formel = Volumen ~ Højde + Omkrets)
koefficienter: (Aflytning) Højde Omkrans -57, 9877 0, 3393 4, 7082 Grad of Freedom: 30 Total (dvs. Nul); 28 Rest Nul afvigelse: 8106 Restafvigelse: 421.9 AIC: 176.9 |
summary(x)
| Opkald:
glm (formel = Volumen ~ Højde + Omkrets) Afvigelsesrester: Min 1Q Median 3Q Max -6.4065 -2.6493 -0.2876 2.2003 8.4847 koefficienter: Estimate Std. Fejl t-værdi Pr (> | t |) (Aflytte) -57.9877 8.6382 -6.713 2.75e-07 *** Højde 0.3393 0.1302 2.607 0.0145 * Omkrets 4.7082 0.2643 17.816 <2e-16 *** - Signif. koder: 0 '***' 0, 001 '**' 0, 01 '*' 0, 05 '.' 0, 1 '' 1 (Dispersionsparameter for gaussisk familie antages at være 15.06862) Nul afvigelse: 8106.08 på 30 frihedsgrader Restafvigelse: 421, 92 på 28 frihedsgrader AIC: 176, 91 Antal Fisher-scoringer: 2 |
Outputet fra opsummeringsfunktionen giver opkald, koefficienter og rester. Ovenstående svar viser, at både højde og omkrets er effektiv, da den er mindre end 0, 5. Og der er to varianter af afvigelse kaldet null og rest. Endelig er fisherscoring en algoritme, der løser problemer med størst sandsynlighed. Med binomial er responsen en vektor eller matrix. cbind () bruges til at binde søjlevektorerne i en matrix. Og for at få detaljerede oplysninger om fit-resuméet bruges.
For at gøre Som hætte test udføres følgende kode.
step(x, test="LRT")
Start: AIC=176.91
Volume ~ Height + Girth
Df Deviance AIC scaled dev. Pr(>Chi)
421.9 176.91
- Height 1 524.3 181.65 6.735 0.009455 **
- Girth 1 5204.9 252.80 77.889 < 2.2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Call: glm(formula = Volume ~ Height + Girth)
Coefficients:
(Intercept) Height Girth
-57.9877 0.3393 4.7082
Degrees of Freedom: 30 Total (ie Null); 28 Residual
Null Deviance: 8106
Residual Deviance: 421.9 AIC: 176.9
Model pasform
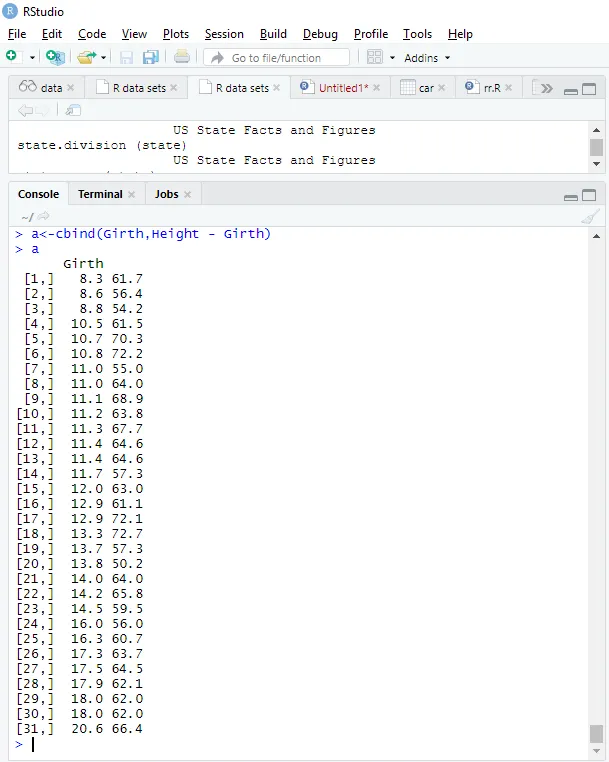
a<-cbind(Height, Girth - Height)
> a
Resumé (træer)
Girth Height Volume
Min. : 8.30 Min. :63 Min. :10.20
1st Qu.:11.05 1st Qu.:72 1st Qu.:19.40
Median :12.90 Median :76 Median :24.20
Mean :13.25 Mean :76 Mean :30.17
3rd Qu.:15.25 3rd Qu.:80 3rd Qu.:37.30
Max. :20.60 Max. :87 Max. :77.00
For at få den rette standardafvigelse
apply(trees, sd)
Girth Height Volume
3.138139 6.371813 16.437846
predict <- predict(logit, data_test, type = 'response')
Dernæst henviser vi til tælleresponsvariablen til modelleret en god responspasning. For at beregne dette bruger vi USAccDeath datasættet.
Lad os indtaste de følgende uddrag i R-konsollen og se, hvordan årtællingen og årskvadratet udføres på dem.
data("USAccDeaths")
force(USAccDeaths)
// At analysere året fra 1973-1978.
disc <- data.frame(count=as.numeric(USAccDeaths), year=seq(0, (length(USAccDeaths)-1), 1)))
yearSqr=disc$year^2
a1 <- glm(count~year+yearSqr, family="poisson", data=disc)
summary(a1)
| Opkald:
glm (formel = antal ~ år + årSqr, familie = “poisson”, data = disk) Afvigelsesrester: Min 1Q Median 3Q Max -22.4344 -6.4401 -0.0981 6.0508 21.4578 koefficienter: Estimate Std. Fejl z-værdi Pr (> | z |) (Opfangning) 9.187e + 00 3.557e-03 2582.49 <2e-16 *** år -7.207e-03 2.354e-04 -30.62 <2e-16 *** årSqr 8.841e-05 3.221e-06 27.45 <2e-16 *** - Signif. koder: 0 '***' 0, 001 '**' 0, 01 '*' 0, 05 '.' 0, 1 '' 1 (Spredningsparameter for Poisson-familie anses for at være 1) Nul afvigelse: 7357, 4 på 71 frihedsgrader Restafvigelse: 6358, 0 på 69 frihedsgrader AIC: 7149, 8 Antal Fisher-scoringer: 4 |
For at kontrollere, om modellen passer bedst, kan følgende kommando bruges til at finde
resterne til testen. Fra nedenstående resultat er værdien 0.
1 - pchisq(deviance(a1), df.residual(a1))
Brug af QuasiPoisson-familien til den større variation i de givne data
a2 <- glm(count~year+yearSqr, family="quasipoisson", data=disc)
summary(a2)
| Opkald:
glm (formel = antal ~ år + årSqr, familie = “quasipoisson”, data = disk) Afvigelsesrester: Min 1Q Median 3Q Max -22.4344 -6.4401 -0.0981 6.0508 21.4578 koefficienter: Estimate Std. Fejl t-værdi Pr (> | t |) (Opfangning) 9.187e + 00 3.417e-02 268.822 <2e-16 *** år -7.207e-03 2.261e-03 -3.188 0.00216 ** årSqr 8.841e-05 3.095e-05 2.857 0.00565 ** - (Dispersionsparameter for quasipoisson-familie anses for at være 92.28857) Nul afvigelse: 7357, 4 på 71 frihedsgrader Restafvigelse: 6358, 0 på 69 frihedsgrader AIC: NA Antal Fisher-scoringer: 4 |
Sammenligning af Poisson med binomial AIC-værdi afviger markant. De kan analyseres ved hjælp af præcisions- og tilbagekaldelsesforhold. Det næste trin er at verificere resterende varians er proportional med gennemsnittet. Derefter kan vi plot med ROCR-bibliotek for at forbedre modellen.
Konklusion
Derfor har vi fokuseret på speciel model kaldet generaliseret lineær model, som hjælper med at fokusere og estimere modelparametrene. Det er primært potentialet for en kontinuerlig responsvariabel. Og vi har set, hvordan glm passer til en R indbyggede pakker. De er de mest populære tilgange til måling af tælledata og et robust værktøj til klassificeringsteknikker, der bruges af en dataforsker. R-sprog hjælper selvfølgelig med at udføre komplicerede matematiske funktioner
Anbefalede artikler
Dette er en guide til GLM i R. Her diskuterer vi GLM-funktionen og hvordan man opretter GLM i R med trædatasæt eksempler og output. Du kan også se på den følgende artikel for at lære mere -
- R Programmeringssprog
- Big Data Arkitektur
- Logistisk regression i R
- Big Data Analytics-job
- Poisson-regression i R | Implementering af Poisson-regression